Fazia poucos dias que um novo exame havia entrado na rotina do Hospital Israelita Albert Einstein quando os profissionais de análises clínicas receberam uma amostra de sangue de um paciente vindo do Hospital das Clínicas da Faculdade de Medicina da USP, em São Paulo. O homem, de 53 anos, apresentava febre, icterícia e olhos avermelhados, sintomas típicos da febre amarela.
Os médicos que o atenderam já haviam realizado exames para investigar, além da febre amarela, dengue, chikungunya e zika, e todos deram negativos. A equipe decidiu, então, recorrer ao Albert Einstein para que uma análise mais minuciosa fosse feita.
Infelizmente ele não resistiu, mas o resultado que saiu dias depois da morte dele, em 11 de janeiro, concluiu que a febre hemorrágica foi causada pelo arenavírus. “Foi uma surpresa para nós”, relata Deyvid Amgarten, aluno de doutorado do Programa Interunidades de Pós-Graduação em Bioinformática da USP e um dos criadores do teste. “Não havia registros da doença no Brasil desde 1990.”
Ainda não se sabe como o homem, que morava em Eldorado, município localizado ao sul do Estado de São Paulo, se infectou. Ele teria viajado para outras três cidades antes de apresentar os primeiros sintomas, em dezembro de 2019.
Análise metagenômica
Viroma é o nome que a equipe do Hospital Israelita Albert Einstein – da qual Amgarten faz parte – deu ao teste genético. Ele é inédito e analisa o material genético de todos os patógenos presentes no organismo de um paciente, incluindo os vírus. Metagenômica é a técnica que permite obter dados de microrganismos de um determinado habitat sem a necessidade de isolar e cultivar cada um separadamente.
O padrão preconizado em laboratórios e hospitais para diagnóstico de doenças infecciosas é a realização de um exame chamado PCR (Reação em Cadeia da Polimerase), que também faz uma análise do genoma de vírus e bactérias. ” A diferença é que ele é específico e direcionado. “Se há uma suspeita de febre amarela, faz-se um PCR para investigar somente essa patologia. Se o exame der negativo, o médico solicita outro exame, e assim sucessivamente, até descobrir a causa”, explica o bioinformata.
O viroma funciona assim: com a amostra de sangue em mãos, faz-se uma separação do material genético do paciente e dos patógenos presentes no material. Extrai-se o RNA e separa-se o que pertence ao vírus e o que é do hospedeiro humano. O RNA é sequenciado e comparado aos vírus catalogados em diferentes bancos de dados, até que seja identificado de qual se trata.
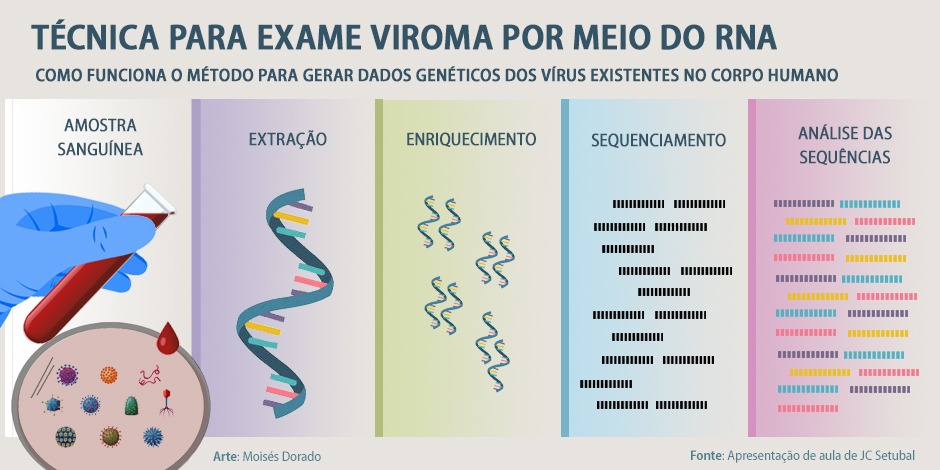
Várias etapas, diferentes desafios
Para que um exame como esse entre em operação, são necessários vários estudos e diferentes testes. No caso do viroma, após um ano de trabalho, ele foi liberado. “Todas as etapas foram desenvolvidas aqui no Brasil e eu fui o responsável pela bioinformática do projeto”, comemora Amgarten.
O primeiro grande desafio que o cientista e a equipe encontraram foi a extração do RNA. “Quando ocorre a separação [do RNA viral e do RNA do hospedeiro], a quantidade de material do vírus que sobra é muito pequena”, explica. “Conseguimos uma maneira de ‘enriquecer’ esse RNA, quer dizer, aumentamos bastante a quantidade de vírus nesse RNA e, assim, conseguimos trabalhar.”
Amgarten foi o responsável pela etapa seguinte – o desenvolvimento de um pipeline, como os profissionais da área chamam. “Pipeline é uma sequência de análises necessárias para produzir um resultado final”, explica o cientista. “A criação desses fluxos é importante porque padroniza os passos de análises e permite gerar o resultado com consistência e confiabilidade”, diz. “Quando olhamos para as sequências dos genomas, elas nem sempre estão completas. Temos que comparar com vários bancos de dados, e isso é trabalhoso, porque há muita informação disponível”. Segundo ele, a padronização permite otimizar os dados para que uma busca seja executada em um tempo razoável e preciso.
A validação do exame foi feita após a realização de testes em 49 amostras. “Passamos pelas etapas de acurácia, reprodutibilidade e sensibilidade. Já sabíamos quais patógenos estavam presentes em cada uma das amostras e comparamos com os resultados gerados pelo viroma”, explica Amgarten. “Todos foram concordantes.”
Expertise formada na Universidade
Amgarten está em contato com a ciência metagenômica desde que iniciou seu mestrado no Laboratório de Bioinformática do Instituto de Química (IQ) da USP, coordenado pelo professor João Carlos Setubal, que atualmente é seu orientador no doutorado. Um dos projetos temáticos em andamento na época era o estudo de comunidades microbianas do Zoológico de São Paulo. “Pegávamos amostras de compostagem, de fezes de macacos bugios e da água dos lagos, extraíamos o DNA e determinávamos quais bactérias estavam ali”, explica Setubal. “Mas quando Deyvid entrou aqui, deixou muito claro que o interesse dele era trabalhar com vírus e bacteriófagos”. Bacteriófagos são vírus capazes de infectar bactérias. Amgarten desenvolveu, então, toda a parte de bioinformática de vírus da pesquisa.
“No laboratório, ele teve contato e acompanhou a execução de diversas metodologias de biologia molecular, incluindo purificação de DNA e RNA a partir de amostras complexas como solo e compostagem, PCR e sequenciamento de DNA de alto desempenho”, explica Aline Maria da Silva, professora associada do Departamento de Bioquímica do Instituto de Química (IQ) da USP e co-orientadora de Amgarten. Ela diz ainda que o fato do cientista vivenciar as rotinas dos laboratórios de Microbiologia e Biologia Molecular lhe deu uma visão de como são gerados dados com qualidade e precisão. “Deyid participou de discussões sobre temas da área que certamente o ajudaram a desenvolver o viroma.”
Deyvid Amgarten – Foto: Arquivo Pessoal
A doença
O arenavírus é um tipo de febre hemorrágica rara, mas de alta letalidade. O período de incubação dura de 7 a 21 dias e os principais sintomas são febre, mal-estar, manchas vermelhas pelo corpo, dor de cabeça, dor atrás dos olhos, além de sangramento de mucosas como boca e nariz. Depois de quatro ou cinco dias, os sintomas pioram: o paciente fica prostrado e pode evoluir para convulsões e coma.
A família dos arenovírus se divide em três: mammarenavírus (infecta mamíferos), reptarenavírus e hartmanivírus (répteis). O arenavírus brasileiro foi identificado pela primeira vez nos anos de 1990 e leva o nome de Sabiá, bairro da cidade de Cotia, em São Paulo, onde foi descoberto. O tipo que apareceu agora tem 88% de similaridade genética com o Sabiá. “De fato, ele é bem similar, mas com apenas um paciente infectado, não há como saber ainda se é um vírus mais ou menos agressivo”, explica Camila Romano, virologista do Instituto de Medicina Tropical (IMT) da USP. “Em geral, a mortalidade dos vírus desse mesmo grupo gira em torno de 30%, quando o tratamento é iniciado tardiamente”.
A transmissão acontece pela inalação de pequenas partículas de urina, fezes ou saliva de roedores silvestres suspensas no ar, além de mordidas de animais. O contágio também pode ocorrer quando há contato muito próximo com a pessoa infectada ou com fluidos corporais, como urina, fezes, saliva, vômito, sangue e sêmen. “A principal forma de prevenção é evitar contato com excrementos de roedores, uma vez que é este o modo natural de transmissão”, alerta a virologista. “Quanto à possibilidade de surto, é improvável”, completa.
Os arenavírus já causaram doenças semelhantes nas Américas e na África.
Com informações de Jornal da USP via Fabiana Mariz.







