As técnicas que utilizam beads magnéticas (SPRI – Solid Phase Resersible Immobilization) para purificação de produtos de PCR (Polymerase Chain Reaction) e amostras de DNA/RNA são amplamente utilizadas em aplicações como sequenciamento pelo método de Sanger ou sequenciamento de nova geração (NGS), genotipagem por SNP (Single Nucleotide Polymorphism), clonagem, análises de fragmentos e primer walking. Com o PIPETMAX® (Fig.1) é possível automatizar de forma fácil, flexível e confiável os protocolos de preparação de bibliotecas e purificação de produtos de PCR por beads magnéticas além da normatização de amostras*.

Fig 1: Pipetmax
A Gilson desenvolveu uma estante separadora de beads magnéticas para uso no PIPETMAX® (Fig. 2), capaz de fazer de forma fácil e prática a purificação de 96 amostras simultaneamente. O protocolo automatizado é flexível, pois pode ser ajustado às variações de corrida para corrida como: número de amostras, volume de cada amostra, volume dos beads, número de lavagens e tempo de incubação. Isso torna o PIPETMAX® a mais simples e flexível plataforma de automação, adequado para inúmeras aplicações.

Fig 2: Estante separadora de beads magnéticas. A base magnética com 24 imãs se eleva para melhor contato com a placa de 96 poços. Cada imã é posicionado entre 4 poços puxando as beads para o respectivo canto do poço.
A purificação de produtos de PCR (amplicons) é necessária para remover primer, nucleotídeos não ligados, sais e enzimas usados na amplificação do DNA e que são considerados contaminantes nas aplicações subsequentes.
Existem várias formas de purificar os produtos de PCR incluindo uso de géis de agarose, colunas de sílica, ponteiras funcionais, digestão enzimática e beads magnéticas. Cada abordagem tem suas vantagens de desvantagens, como custo, rapidez e qualidade. As beads magnéticas, por sua vez, são as mais difundidas e comumente usadas nas aplicações de NGS, por concentrarem o maior número de vantagens em um único método são facilmente automatizadas, rapidamente escaláveis e com melhor custo. A Illumina, e também outros fornecedores de soluções para NGS, Thermo, Roche, Sigma- KAPA, New England Biolabs e TAKARA, recomendam especificamente o uso de kits de purificação de PCR por beads como o Agencourt® AMPure XP para a etapa de limpeza – clean up no fluxo de trabalho da preparação de bibliotecas NGS.
MATERIAL e MÉTODO
Foram geradas 12 bibliotecas a partir de DNA genômico de Escherichia coli K12 para sequenciamento completo do genoma usando o kit Nextera® XT DNA Library Preparation, usando 12 diferentes pares de primers específicos para indexação das amostras (1 ng a 0,2 ng/µl). Como parte do processo, o kit Agencourt® AMPure XP PCR Purification foi automatizado no PIPETMAX® visando a purificação dos produtos de PCR.
A configuração utilizada no PIPETMAX® utilizou o pipetador multicanal de 8 canais, placas de 96 poços, ponteiras Gilson DIAMOND®, tubos de PCR em estantes, reservatórios de descarte de ponteiras e estante magnética para separação das beads.
O micro software TRILUTION® foi usado para a programação do PIPETMAX®. Sua interface gráfica intuitiva guia facilmente o usuário para a criação do protocolo.
As mesmas 12 bibliotecas foram geradas manualmente para comparação dos resultados.
RESULTADOS
Após a purificação dos produtos de PCR, os fragmentos de DNA foram avaliados com relação a qualidade e robustez do método por meio de eletroforese analítica em chip 2100 usando o BioAnalyser da Agilent. Abaixo pode-se conferir o eletroferograma das amostras (Fig 3). A maioria dos fragmentos tinha 700-2000 pares de bases sem presença de contaminantes.
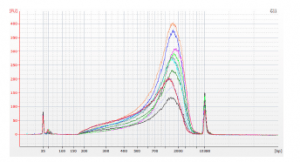
Fig.3 Eletroferograma de fragmentos gDNA de E. coli, após processamento com o kit Nextera® XT DNA Library Preparation e purificação usando o Agencourt® AMPure XP PCR Purification no PIPETMAX® e manualmente.
As 24 bibliotecas foram agrupadas para sequenciamento conjunto no sistema Illumina MiSeq®.
Os resultados mostraram que a porcentagem de leituras mapeadas foi praticamente idêntica tanto nas bibliotecas preparadas manualmente quanto no PIPETMAX® (Tabela 1). De maneira geral, as bibliotecas preparadas no PIPETMAX® apresentaram valores menores de desvios padrão e variâncias do que aquelas preparadas manualmente (vide Tabela 1). A métrica de qualidade, %>Q30, foi melhor para amostras processadas no PIPETMAX®, quando comparado com as processadas manualmente. Essa análise comparativa indica que o PIPETMAX® gera resultados mais uniformes que os obtidos manualmente. Ambas preparações, manual ou automatizada tiveram mais de 95% de leituras mapeadas.
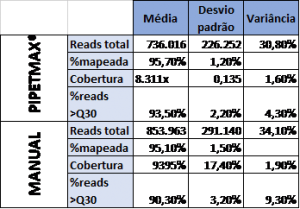
Tabela 1: resumos dos resultados do sequenciamento
CONCLUSÃO: Todos os resultados obtidos indicam que o PIPETMAX® é adequado e reprodutível para preparação de amostras para NGS.
*Consulte www.gilson.com para saber mais sobre o protocolo de normalização de amostra de DNA/RNA).
REFERÊNCIAS:
- PIPETMAX®: Automation of the Illumina Nextera® XT DNA Library Preparation kit, Application Note AN1011 Acessado em abril, 2020: https://www.gilson.com/pub/static/frontend/Gilson/customtheme/en_US/images/docs/PIPETMAX_NexteraDNALibrary_AN1011-01.pdf)
- PIPETMAX®: Automation of Agencourt AMPure XP PCR Prification System, Application Note AN1010 Acessado em abril, 2020: https://www.gilson.com/pub/static/frontend/Gilson/customtheme/en_US/images/docs/PIPETMAX_AMPureXPPCR_AN1010.pdf






